Kontrolowana ewolucja w probówce. Pierwszy taki enzym
Stworzono już mnóstwo enzymów, które zaprojektowano komputerowo i zsyntetyzowano w laboratorium. Nikomu wcześniej nie udało się jednak odtworzyć ewolucji enzymu w probówce. Aż do teraz.
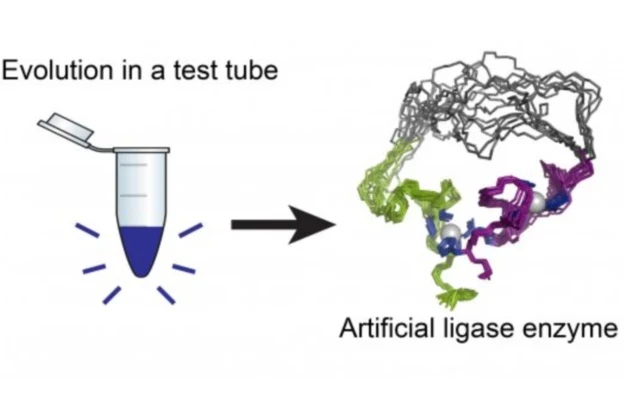
Tradycyjne metody projektowania enzymów (substancji biologicznych przyspieszających reakcje chemiczne) zakładają stworzenie komputerowego modelu cząsteczki, zanim rozpocznie się ich produkcję w laboratorium. Takie podejście pozwala na modyfikację właściwości chemicznych danego enzymu, jeszcze zanim ten powstanie.Naukowcy z University of Minnesota spróbowali innego podejścia, dzięki czemu otrzymali pierwszą syntetyczną, "żelkową ligazę". Jak opisują sami odkrywcy, stworzony przez nich katalizator to bardzo dynamiczny, drgający enzym, który przeprowadza reakcję ligacji cząsteczek RNA.Do stworzenia opisywanego enzymu naukowcy wykorzystali nie powszechnie wykorzystywane algorytmy bioinformatyczne, a losowe kreowanie łańcuchów aminokwasowych oraz manualną selekcję tych, które spełniają założone przez naukowców kryteria. Proces ten jest o wiele bliższy ewolucji, niżeli powszechnie stosowane metody.Koordynator projektu - Burckhard Seelig - przekonuje, że dzięki zastosowanej metodzie, jego zespołu nie ograniczają sztywne reguły stosowane podczas projektowania białek in silico (przy użyciu komputera). Dzięki temu możliwe jest losowe tworzenie substancji, które wykazują aktywność enzymatyczną. Stworzona przez uczonych z University of Minesota ligaza RNA jest bardzo pierwotnym, niezbyt wydajnym enzymem. Tak właśnie mogły wyglądać prehistoryczne biologiczne katalizatory, u organizmów żywych na wczesnych etapach ewolucji.










